FINANCEMENT : ANR Appel à projets générique 2021
AAP Mathématiques et sciences du numérique pour la biologie et la santé.
Porteur : Eric HERBERT (LIED UMR 8236)
Coordinateurs scientifiques : F. CHAPELAND-LECLERC (LIED UMR 8236), A. VEBER (MAP5 UMR 8145) et Y. D’ANGELO (LJAD UMR 7351)
2021-2025. 367 000 €.
Autres financements obtenus sur cette thématique :
-IDEX Emergence en recherche HWW (2019-2021)
-CNRS MITI Défi Modélisation du Vivant « DREAMS » (2019 et 2020) /workshop DREAMS (porteur Y. D’Angelo)
-CNRS INSIS Ingénierie inspirée par la Nature « PhysNet » (2020-2021) (porteur M. Durand)
DESCRIPTIF
Comment des champignons ou des plantes envahissent ils un milieu ? comment des maladies se propagent au sein d’une population ? comment les voies de communication se densifient ? Plus généralement, comment l’information, la masse, l’énergie se propagent dans un réseau ? Ce sont des questions qui semblent s’adresser à des problèmes a priori sans rapport. En réalité, la structure, la dynamique et la forme du réseau sous-jacent peuvent être décrits par des modèles très similaires, allant de modèles explicatifs simples [Albin et d’Angelo, 2012 ; Heaton et al., 2012] à des approches plus réalistes, ces modèles dépendent des caractéristiques locales d’un grand nombre, éventuellement croissant, de connections entre des nœuds, évoluant dans le temps et de leurs interactions dans des systèmes étendus.
Le thalle des champignons filamenteux est un cas intéressant de réseau branchant en croissance. Il est constitué d’un réseau d’hyphes interconnectées, le mycélium, qui permet une exploration spatiale efficace des ressources nutritives. In vivo, la croissance du réseau fongique doit faire face à contraintes externes qui perturbent ou empêchent l’exploration de l’environnement [Fricker et al., 2017]. Notons que la question des échelles est ici primordiale : l’hyphe a une largeur de quelques microns (typiquement 4 à 6), alors que le réseau mycélien peut opérer à des échelles spatiales allant de quelques millimètres à plusieurs kilomètres [Lew, 2011].
Dans ce projet, nous abordons spécifiquement la caractérisation de la croissance du réseau fongique du champignon filamenteux Podospora anserina [Silar, 2020]. Au-delà de l’intérêt fondamental de mieux comprendre comment ce champignon filamenteux s’adapte à un environnement compétitif, il représente un bon modèle de laboratoire pour étudier le développement des champignons filamenteux, et plus généralement, des réseaux d’autres micro-organismes. Nous souhaitons aborder le problème de la modélisation et de l’analyse multi-échelle de réseaux dynamiques en expansion sous des contraintes externes, à la fois par des moyens analytiques/numériques et par des réalisations expérimentales à l’échelle du laboratoire. Afin d’acquérir et d’analyser une quantité suffisamment importante d’ensembles de données, à la fois à petite et grande échelle, les expériences suivantes sont entreprises :
1/ Développement d’une description quantitative du réseau d’hyphes en se positionnant au niveau cellulaire par la mise en place d’un système optique de fluorescence, en utilisant des conditions et des contraintes faciles à mettre en œuvre et en caractérisant certains mutants affectés dans le développement du réseau fongique.
2/ Développement d’une procédure de routine pour effectuer une reconstruction et un prétraitement efficaces des images. L’objectif est de transformer les données expérimentales brutes en un graphe spatio-temporel codant la dynamique du réseau avec un étiquetage efficace des apex, et capable de discriminer les anastomoses (fusion) des nœuds (intersection). La combinaison des traitements classiques d’images à des post-traitements géomatiques (ces derniers étant habituellement réalisés en sciences humaines et environnementales) constitue l’une des originalités de ce projet.
3/ Développement de la modélisation et des simulations numériques en deux étapes. A l’échelle du réseau, en modélisant et en étudiant les statistiques clés de la structure du réseau (fréquence, distribution spatiale, distribution angulaire des points de branchement, vitesse des apex…), et à l’échelle moléculaire, en utilisant des outils stochastiques et déterministes pour construire un modèle de la dynamique de concentration des protéines déclenchant la croissance et le branchement des hyphes.
Récemment, nous avons développé un dispositif expérimental automatisé et reproductible pour suivre la croissance du réseau d’hyphes de P. anserina [Dikec et al., 2020]. Le réseau fongique est ici considéré comme une entité unique, définie par ses caractéristiques macroscopiques et comme un assemblage d’hyphes interconnectés à l’échelle microscopique (Fig. 1). Nous avons choisi de nous concentrer sur la croissance du réseau en deux dimensions, en lumière blanche, en conditions standard de culture. Cela nous a permis d’obtenir des séries temporelles d’images du thalle fongique, suivi par un traitement ad-hoc des images. Nous avons ainsi été en mesure d’extraire (i) les quantités globales marquant la croissance du réseau (i.e. la longueur de la matière vivante (L)), (ii) sa complexité, représentée par le nombre de nœuds (N) et le nombre d’apex (A), et (iii) la distribution de la surface intra-thalle (S). Cette exploration spatiale et temporelle systématique nous a permis d’estimer un ensemble de caractéristiques physiologiques clés du réseau fongique, telles que le taux d’anastomose, la dynamique de branchement et la structuration spatio-temporelle du réseau. Toutes ces quantités sont d’une importance cruciale pour guider et évaluer la modélisation, en particulier pour identifier certains paramètres requis dans la simulation à l’échelle micro et macro.
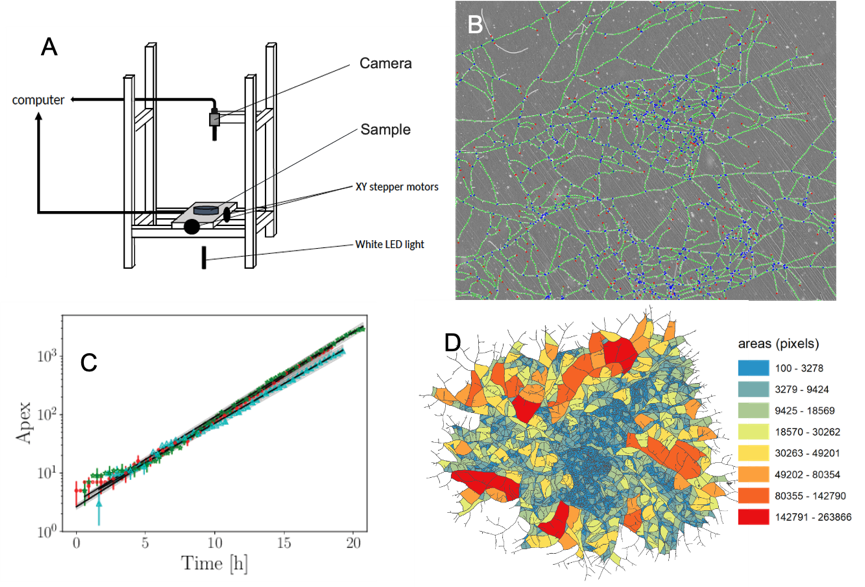
Figure 1. A. Schematic representation of the experimental set-up. B. Vectorized picture of the growing hyphal network of P. anserina, apexes in red, nodes in blue and hyphae in green from the bright led microscopy setup. C. Evolution of number of apexes in function of time. D. Distribution of intra-thallus areas.
Bibliographie
1-E. Albin and Y. D’Angelo. In: Combustion and Flame 159.5 (May 1, 2012), pp. 1932-1948.
3-J. Dikec et al. In: Scientific Reports 10.1 (Feb. 21, 2020), pp. 1-16.
4-M. D. Fricker et al. In: Microbiology Spectrum 5.3 (2017).
5-L. L. M. Heaton et al. In: Physical Review. E, Statistical, Nonlinear, and Soft Matter Physics 86.2 (2012), p. 021905.
6-R. R. Lew. In: Nature Reviews. Microbiology 9.7 (June 6, 2011), pp. 509-518.
7-P. Silar. HAL, Feb. 2020.
PERSONNES ET LABORATOIRES IMPLIQUES :
– LIED UMR 8236 (Université de Paris), physique et géomatique : Eric HERBERT, Pascal DAVID, Christophe LALANNE, Cécilia BOBEE et Frédéric FILAINE.
– LIED UMR 8236 (Université de Paris), biologie : Florence CHAPELAND-LECLERC, Gwenaël RUPRICH-ROBERT et Eva CABET.
– LJAD UMR 7351 (Université Nice Sophia Antipolis/Université Côte d’Azur), modélisation : Yves D’ANGELO, Rémi CATELLIER, Laurent MONASSE et Claire GUERRIER.
– MAP5 UMR 8145 (Université de Paris), probabilités : Amandine VEBER et Milica TOMASEVIC.
Deux doctorants ont été recrutés sur ce projet : Clara LEDOUX (bourse doctorale MTCI 2019-2023) et Thibault CHASSEREAU (financement ANR, 2021-2024).